- I) Généralités
- II) Transcription de l’ADN procaryote
- 1) Pré-inscription
- 2) Initiation
- 3) Elongation
- 4) Terminaison
- 5) Maturation des transcrits primaires
- III) Transcription de l’ADN eucaryote
- 1) Les ARN-polymérases eucaryotes
- 2) Différence dans la transcription eucaryote
- a) Complexe protéique nécessaire à la transcription
- b) Promoteur minimum et régions régulatrices
- 3) Les régions cis-régulatrices
- a) Les séquences amplificatrices de types enhancers
- b) Les séquences extinctrices de types silencers
- c) Les séquences isolantes de types insulators
- 4) Caractéristiques structurales des protéines régulatrices
- 5) Maturations des transcrits primaires
- a) Addition de la coiffe en 5′ (ou capping)
- b) Poly-adénilation en 3′ par la poly-A polymérase
- c) Excision des introns et épissage des exons (ou splicing)
- d) Epissage alternatif
I) Généralités
Le gène (ou cistron) est un segment d’ADN qui constitue l’unité d’expression menant à la formation d’un produit fonctionnel qui peut être sous la forme d’ARN ou de polypeptide.
Chez les procaryotes plusieurs gènes peuvent faire partie d’une même unité de transcription, on parle d’unité polycistronique dont l’exemple le plus classique est l’opéron lactose (cf. chapitre de régulation de l’expression des gènes).
Chez les eucaryotes les unités de transcription sont monocistronique (exception chez certains vertébrés).
Les gènes eucaryotes sont départagés dans 3 classes :
- Les gènes de classe 1 ont comme produits des ARNr. Ils sont répétés en tandem et séparés par des espaces inter-géniques. La transcription des ARNr se fait sous forme d’un précurseur l’ARN 45 S (≈ 13 000 pdb) qui sera clivé en 3 ARNr : 5,8 S ; 18 S et 28 S.
- Les gènes de classe 2 ont comme produits des protéines. Les protéines sont formées par traduction des ARNm qui sont plus instables que les autres ARN. Les gènes de classe 2 sont constitués de différents segments : des exons portant l’information et correspondant aux régions codantes, et des introns qui sont des régions non codantes. Attention, les procaryotes ne possèdent pas d’introns.
- Les gènes de classe 3 ont comme produits des ARNt, les ARNr 5S et les petits ARN qui sont également répétés en tandem comme les gènes de classe 1.La synthèse d’ARN à lieu essentiellement pendant l’interphase du cycle cellulaire.
II) Transcription de l’ADN procaryote
L’ARN polymérase est une protéine ADN dépendante, multimérique possédant les sous-unités α, β, β’ et σ. Elle est présente sous deux formes l’enzyme-cœur (α2ββ’) et l’holoenzyme (α2ββ’σ). Les ARN polymérases ne nécessitent pas d’amorce et ne possèdent pas d’activité exo-nucléasique et donc de correction d’erreur, le taux d’erreur est ainsi plus important que pour les ADN-polymérases, mais ce taux est supporté.
Les nucléotides triphosphates sont additionnés à l’extrémité 3’ de la chaîne en cours de synthèse par complémentarité de la matrice d’ADN. L’hydrolyse de la liaison anhydride fournit l’énergie pour la synthèse de la liaison phosphodiester.
La réaction est réalisée en milieu tamponné à pH neutre, contenant du sel de Mg2+, un agent de protection des groupements SH de l’enzyme (agent réducteur comme le β-mercaptoéthanol), les 4 ribonucléotides (rNTP) et de l’ADN bicaténaire contenant un promoteur comme matrice.
La molécule d’ADN est composée d’un brin matriciel (ou brin anti-codant) dirigé par définition de 3’ vers 5’ et servant comme son nom l’indique de matrice à l’ARN-polymérase, ainsi que d’un brin codant dirigé par définition de 5’ vers 3’ et ayant une séquence identique à l’ARN transcrit mise à part le fait que la thymine est changée par l’uridine. L’ARN messager est lui monocaténaire et dirigé tout comme le brin codant de 5’ vers 3’.
Chez E-coli, une seule ARN-polymérase catalyse la synthèse de tous les ARN de la cellule (en mettant à part l’ARN des amorces nécessaire à la réplication de l’ADN).
La transcription est divisée en plusieurs étapes : la pré-initiation, l’initiation, l’élongation et la terminaison.
1) Pré-initiation
Le promoteur est une séquence d’une centaine de nucléotides située dans la région régulatrice et désignant le début de la transcription. Il est situé en amont du site d’initiation et porte des éléments de séquence reconnus par l’ARN-polymérase et déterminant le sens de la transcription.
Le promoteur est constitué de courtes séquences conservées d’une unité de transcription à l’autre et appelées séquences consensus :
- En -10 du site d’initiation on trouve la TATA box ou boîte de Pribnow : « TATAAT »
- En -35 du site d’initiation on trouve : « TTGACA »
La force (ou efficacité) intrinsèque d’un promoteur est définie par le nombre relatif d’initiation par unité de temps (vitesse de la transcription). Elle dépend de la proportion des paires de bases A-T par rapport aux paires de bases C-G, de la position des séquences en -35 et en -10, en effet plus les séquences consensus sont proches du site d’initiation plus le promoteur sera fort et finalement de la force d’interaction de l’ARN-polymérase avec le promoteur.
Le promoteur agit sur la transcription du segment d’ADN qui lui est adjacent sur le même chromosome, on dit que le promoteur est actif en « cis ». Le promoteur n’est pas actif sur les séquences codantes situées ailleurs sur le chromosome, dans ce cas là on parlerait alors du qualificatif « trans ». Le cis s’oppose au trans.
L’affinité de l’ARN-polymérase pour l’ADN dépend de la forme de l’enzyme : l’enzyme-cœur a une affinité faible et non spécifique, l’holoenzyme a une affinité très forte et spécifique pour le promoteur. On peut faire la remarque que la sous-unité sigma σ à l’état libre ne se fixe pas sur l’ADN. La sous-unité β’ étant basique et l’ADN étant acide, ce sera elle qui facilitera l’interaction du complexe avec le promoteur.
La sous-unité sigma σ permet donc une reconnaissance spécifique du promoteur par l’ARN-polymérase et diminue l’affinité de l’enzyme pour les régions non promotrices. Il agit de manière cyclique, en effet après l’initiation faite, le facteur sigma se détache pour être recyclé et réutilisé pour d’autres initiations de gènes.
L’ARN-polymérase entraîne la dénaturation des deux brins d’ADN sur 14 paires de nucléotides, on parle de complexe ouvert qui augmente encore l’affinité de l’enzyme pour la double hélice.
2) Initiation
L’initiation correspond à la synthèse de la première liaison phosphodiester réalisé par la sous-unité β qui correspond à la sous-unité catalytique de l’ARN-polymérase.
L’interaction de cette sous-unité est inhibée par la rifampicine qui inhibe ainsi de manière irréversible la transcription de l’ADN, c’est le cas de la tuberculose. Une mutation dans la SU β induit l’apparition de souches bactériennes résistantes à la rifampicine.
Le déroulement des premières étapes de la transcription est donc :
- liaison non spécifique de l’holoenzyme.
- formation d’un complexe fermé au niveau du promoteur
- formation du complexe ouvert (déroulement sur 14 nucléotides)
- Mise en place du premier nucléotide (très souvent A ou G)
- Allongement de 4 à 5 nucléotides
- Détachement du facteur sigma, après la transcription des 4-5 premiers nucléotides.
3) Elongation
L’élongation correspond au déplacement de la bulle de transcription le long de la molécule d’ADN. La région désappariée est alors de 70 paires de bases. Pendant la transcription, l’ARN forme un court appariement avec le brin matriciel de l’ADN formant une hélice hybride ADN-ARN sur une dizaine de paires de bases.
L’élongation est inhibée par des aminosides ou amino-glucosides.
4) Terminaison
La terminaison se fait lorsque l’enzyme arrive au niveau d’une séquence spécifique appelée terminateur.
Le terminateur se présente sous la forme d’un palindrome (cf. lexique) qui peut être parfait ou imparfait. Ce palindrome entraîne une complémentarité de séquence au niveau de l’ARNm qui permet la mise en place d’une structure en épingle à cheveux (ou tige-boucle) qui est un appariement intra-chaîne qui déstabilise l’ARN-polymérase jusqu’à dissociation.
Elle peut être facilitée par un facteur rho ρ suivant la séquence du terminateur, on met ainsi en évidence des terminateurs rho indépendant (environ les 2/3) et des terminateurs rho dépendant (environ 1/3) :
- Pour les terminateurs rho indépendant on trouve une structure en épingle à cheveux riche en paires de bases G-C, suivie d’une séquence poly-U d’environ 6 nucléotides permettant une dissociation plus facile de l’hybride ADN-ARN.
- Pour les terminateurs rho dépendant on trouve une structure en épingle à cheveux plus courte et qui n’est pas riche en paires de bases G-C et qui est non-suivie d’une séquence poly-U. Il y a donc nécessité du facteur rho qui a une affinité pour les ARN en court de synthèse, le parcourant de 5’ vers 3’ jusqu’à trouver l’ARN-polymérase. Le facteur rho est ATP dépendante, dont l’hydrolyse permettra la dissociation du complexe.
5) Maturation des transcrits primaires
Le transcrit primaire correspond à l’ARN non mature qui nécessite une maturation sous forme de clivages ou de modifications de bases. Cette maturation n’est pas obligatoire. Après maturation on obtient l’ARN mature. On peut prendre quelques exemples comme la maturation du transcrit primaire donnant les ARNr (transcrit primaire 45 S) ou les ARNt par des ribonucléases, ou la maturation du transcrit primaire codant les ARNm. Généralement, il y a peu de modifications pour les ARNm ; beaucoup d’ARNm sont traduits en protéines alors qu’ils sont encore transcrits (Attention : il n’y a pas de noyau chez les procaryotes).
Le transcrit primaire code soit pour un produit, on parle d’ARN monocistronique, soit plusieurs produits, on parlera alors d’ARN polycistronique (cf. début du cours, généralités).
Vidéo résumant les différents mécanismes entrant en jeu dans l’épissage.
III) Transcription de l’ADN eucaryote
1) Les ARN-polymérases eucaryotes
Trois ARN-polymérases eucaryote ont été mis en évidence. Elles diffèrent par leur localisation dans le noyau, par la nature des ARN formés et de par leur sensibilité à des inhibiteurs tels que l’α-amanitine.
- ARN-polymérase I dans le nucléole pour les ARNr 5,8 ; 18 et 28 S, et est insensible à l’α-amanitine
- ARN-polymérase II dans le nucléoplasme pour les ARNm et est sensible à l’α-amanitine
- ARN-polymérase III dans le nucléoplasme pour les ARNt, ARNr 5 S et pour les petits ARN, elle est également sensible à l’α-amanitine mais à hautes doses.
L’α-amanitine se fixe sur certaine sous-unité de l’ARN-polymérase et inhibe l’élongation de la transcription.
L’actinomycine D inhibe la transcription eucaryote et procaryote en s’intercalant entre certaine base de l’ADN pendant l’élongation.
2) Différence dans la transcription eucaryote
a) Complexe protéique nécessaire à la transcription
L’ARN-polymérase II n’étant pas suffisante pour démarrer la transcription, elle nécessite d’autres protéines interagissant avec l’ADN du promoteur, appelées TFII (pour transcription factor II, facteurs de transcriptions interagissant avec l’ARN-polymérase II) :
- TFII D interagit avec l’ADN du promoteur et plus spécifiquement à la TATA box lorsqu’elle existe.
- TFII A interagit avec l’ADN en amont de la TATA box.
- TFII B interagit avec l’ADN en aval de la TATA box au niveau du site d’initiation.
- TFII F agit lors de l’élongation.
- TFII H possède une activité hélicase, une activité de réparation de l’ADN dans le système NER et une activité kinase qui sert à phosphoryler l’ARN-polymérase II au niveau de son domaine C-terminal (CTD, pour carboxy-terminal domain) nécessaire à l’activation de la transcription. Une déphosphorylation est nécessaire pour permettre une nouvelle pré-initiation. L’extrémité CTD est formée par un enchaînement de sérine pouvant être phosphorylée.
b) Promoteur minimum et régions régulatrices
Les séquences consensus sont plus nombreuses que chez les procaryotes, on trouve :
- La TATA box (= boîte de Hogness) entre -30 et -25, elle est présente dans environ 80% des promoteurs.
- L’INR box à partir du +1 et présente dans environ 60% des promoteurs.
- La GC box
- La CAAT box
Le promoteur eucaryote est une structure modulaire. La TATA box et à l’INR box forme le promoteur minimum au niveau duquel se fixe l’ARN-polymérase II via les facteurs généraux de transcription.
On trouve en plus des séquences activatrices et amplificatrices jusqu’à -200 en amont du site d’initiation ; parmi elles on trouve la GC box et la CAAT box. Les séquences activatrices sont très variables et sont plus ou moins présentent à des nombres également variables suivant le promoteur. Ces boîtes constituent les sites de fixation des facteurs de transcription et permettent ainsi la modulation de l’activité du promoteur minimum.
Le terminateur au niveau de l’ARN est constitué de la séquence CPSF (AAUAAA) suivie par un site de poly-adénilation 20 nucléotides en aval, elle-même suivie de la séquence CSTF qui est une séquence riche en G/U qui s’apparie avec la séquence CPSF (formation d’une structure tige-boucle) et après laquelle il y aura clivage (cf. maturation du transcrit primaire, suite du cours).
3) Les régions cis-régulatrices
a) Les séquences amplificatrices de type enhancers :
Les enhancers fixent des protéines qui vont permettre l’amplification de l’expression des gènes de 10 à 100 fois. Ils peuvent agir en trans sur un promoteur hétérologue et sont actifs dans les deux directions. Ils sont généralement situés en amont du site d’initiation mais peuvent également se situer en aval au milieu de l’unité de transcription.
On pourra prendre comme exemple les récepteurs des hormones thyroïdiennes et les récepteurs des hormones stéroïdes (glucocorticoïde, progestérone, œstrogène). Les deux sont des récepteurs nucléaires et il y a ainsi action au niveau du noyau par modulation de l’expression de certains gènes.
b) Les séquences extinctrices de type silencers :
Les silencers sont des séquences fixant des protéines qui inhibent l’expression des gènes en agissant à distance.
c) Les séquences isolantes de type insulators :
Les insulators sont des séquences isolantes qui permettent d’isoler certaines régions du génome.
4) Caractéristiques structurales des protéines régulatrices
- Le motif hélice-boucle-hélice : une hélice de liaison à l’ADN au niveau du grand sillon et une hélice de stabilisation reliée par la boucle.
- Les motifs en doigt de zinc sont de plusieurs types, dont celui du type 4 cystéines ou du type 2 cystéines + 2 hydrogènes.
- Les leucines zipper correspondent à des hélices amphipathiques présentant des leucines qui interagissent par des liaisons hydrophobes leucine-leucine.
5) Maturation des transcrits primaires
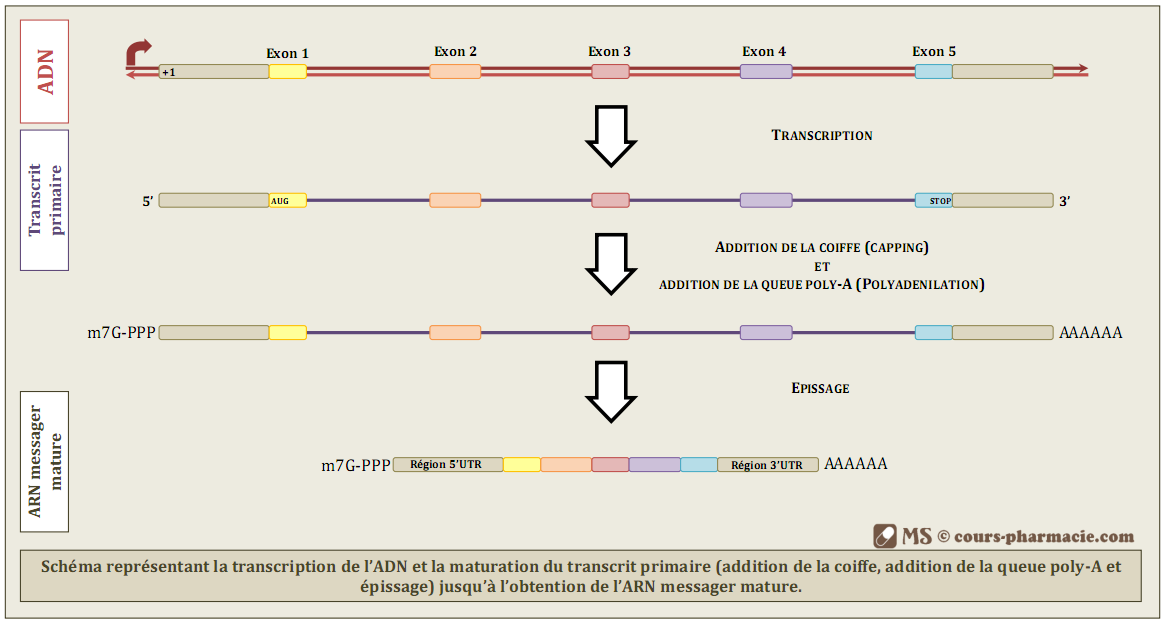
La maturation des transcrits primaires à lieu dans le noyau de la cellule. Chez les procaryotes ce phénomène n’existe pas, le début de la traduction de l’ARNm se faisant avant la fin de la transcription, la cellule procaryote ne possédant pas de noyau.
Les transcrits primaires ne correspondent qu’aux produits de la transcription de l’ARN-polymérase II, ce qui ne veux pas pour autant dire que les produits de la transcription des autres ARN-polymérases ne sont pas soumis à des modifications post-transcriptionnelles. On parle de pré-ARNm, pré-ARNr et pré-ARNt.
a) Addition de la coiffe en 5’ (ou capping)
La coiffe correspond à l’ajout d’un groupement, dit « m7G », par une liaison 5’-5’ tri-phosphate. Ce groupement m7G correspond à l’addition de trois groupements phosphates et d’une molécule de GTP au niveau de l’extrémité 5’ du transcrit primaire grâce à l’énergie libérée par l’hydrolyse de la molécule de GTP. Le nucléotide G va ensuite être méthylé sur le septième carbone (C7) pour donner la 7-méthyl-guanosine.
La coiffe est ajoutée grâce à un complexe protéique appelé « Cap-Binding-Complex » qui possédant une activité triphosphatase, une activité guanylyl-transférase et une activité méthyl-transférase.
b) Poly-adénilation en 3’ par la poly-A polymérase
La poly-adénilation correspond à l’ajout de jusqu’à 200 adénines à l’extrémité 3’ du transcrit primaire et ceci sans matrice par la poly-A-polymérase. La poly-A-polymérase reconnaît le signal de poly-adénilation qui n’est autre que la séquence CPSF (AAUAAA). 20 nucléotides en aval de cette séquence la poly-A-polymérase utilise sont activité endo-nucléasique et son activité A-polymérasique.
c) Excision des introns et épissage des exons (ou splicing)
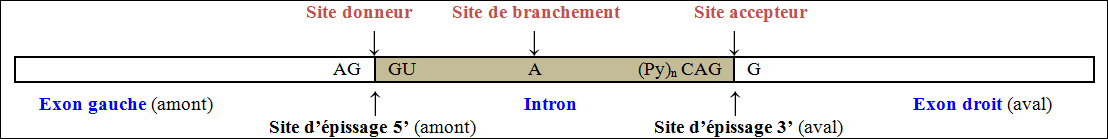
Après l’addition de la coiffe et la poly-adénilation, le transcrit primaire est encore soumis à l’excision des introns et l’épissage des exons ; les introns sont ainsi éliminés. Ceci est possible par la présence de site donneur d’épissage (dinucléotide GU) à l’extrémité 5’ des introns et de site accepteur d’épissage (dinucléotide CAG) à l’extrémité 3’ des introns.
Les jonctions d’épissage sont reconnues par les snRNPs (ou snurps pour Small-Nuclear-Ribonucleo-protein-Particules). Les snRNP correspondent à l’association de snRNA (snRNA U1, U2, U3, U4, U5, U6) et de protéines et l’ensemble des snRNPs s’appelle le spliceosome. Le snRNP U1 reconnaît le site donneur et le snRNP U2 reconnaît le site de branchement et le site accepteur.
L’excision des introns et l’épissage des exons se fait en plusieurs étapes :
- Le snRNP U1 permet la reconnaissance du site donneur d’épissage et entraîne la rupture de la liaison phosphodiester entre le premier exon et l’intron.
- Cette rupture de la liaison phosphodiester entraîne la formation d’un lasso, qui n’est autre que l’extrémité 5’ de l’intron. Ce lasso forme une liaison avec le site de branchement, lui-même situé sur le même intron qui se replit ainsi sur lui-même. Le site de branchement est reconnu par le snRNP U2 et permet la liaison par l’intermédiaire d’une adénosine.
- Le snRNP U2 permet également la reconnaissance du site accepteur d’épissage. Suite à cette reconnaissance il y a rupture de la liaison phosphodiester au niveau de l’extrémité 3’ de l’intron.
- Le groupement 3’OH du premier exon peut ainsi réagir avec l’extrémité 5’phosphate du deuxième exon pour former une liaison phosphodiester et permettre la libération de l’intron qui sera dégradé.
Remarque :
Il existe certains ARN qui possèdent des introns auto-catalytiques qui ne nécessitent ainsi aucune protéine, l’activité enzymatique est portée par l’ARN lui-même, on parle de ribozymes.
Vidéo résumant les différents mécanismes entrant en jeu dans l’épissage.
d) L’épissage alternatif
A partir d’un transcrit primaire on peut avoir deux ou plus ARNm matures qui seront à l’origine de la formation des protéines-isoformes. Ceci est possible grâce à l’épissage alternatif qui consiste en l’élimination de certains exons. En effet certains exons sont constants au niveau des différents ARNm matures et d’autres sont variables et spécifiques du tissu dans lequel se trouve la protéine isoforme.
Exemple :
Au niveau du gène de la tropomyosine on met en évidence 12 exons au total dont 7 sont constants et 5 sont alternatifs.
Pathologies liées à un épissage anormal suite à des mutations :
On prendra pour exemple les β-thalassémies qui correspondent à des anémies héréditaires transmises sur le mode dominant dues à des anomalies dans la production de l’hémoglobine adulte. Certaines mutations induisent un épissage anormal du transcrit primaire (au niveau des sites donneurs, sites de branchement ou sites accepteurs).