- I) Le réplicon
- II) Réplication bidirectionnelle
- III) Polymérisation unidirectionnelle et réplication semi-conservative
- IV) Réplication semi-discontinue
- V) Les ADN polymérases
- 1) Généralités
- 2) Activités des ADN polymérases
- 3) ADN polymérases procaryote dans la réplication
- 4) Le polymorphisme de l’ADN
- VI) Mécanismes de la réplication procaryote chez E-Coli
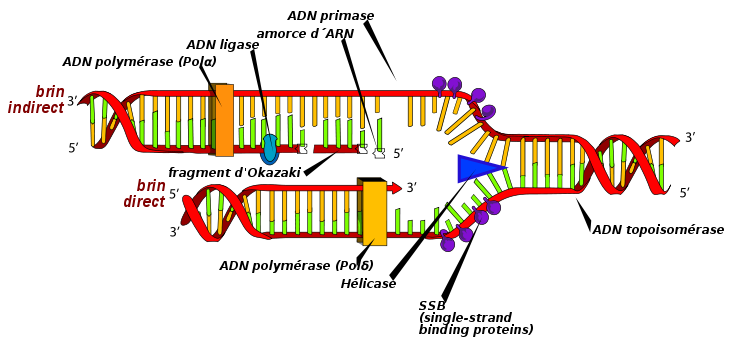
- 1) Les différentes protéines mise en jeu
- 2) Origines et terminaisons chez E-Coli
- 3) Les étapes de la réplication procaryote
- 4) Régulation de la réplication chez E-Coli
- VII) La réplication eucaryote
- 1) Les ADN polymérases eucaryotes
- 2) Les télomères
- VIII) La réplication du matériel génétique des rétrovirus
La phase de réplication est rapide et fiable, et correspond à la reproduction de l’ADN à l’identique. Durant cette phase il peut y avoir des brassages mais aucune information n’est perdue. La réplication s’effectue entre la phase G1 et G2 du cycle cellulaire, c’est la phase « S ».
La réplication de l’ADN doit respecter deux principes :
- l’ensemble du génome doit être répliqué à chaque division cellulaire
- chaque molécule d’ADN n’est répliquée qu’une seule fois par cycle cellulaire.
Exception :
Les chromosomes polythènes sont soumis à des divisions sans mitose (= endomitose) qui entraîne une accumulation de copie d’ADN dans la cellule.
I) Le réplicon
Le réplicon est l’unité de réplication de l’ADN eucaryote, il contient une origine et une terminaison (Remarque : L’ADN procaryote est circulaire et présente une seule origine de réplication). En effet l’ADN peut être répliqué à plusieurs endroits en même temps, sans cela la réplication de l’ADN eucaryote durerait 800 heures et l’homéostasie de l’organisme ne serait pas respectée.
Les réplicons sont des segments de taille variant de 30 000 à 150 000 bases et dont le nombre peut aller de 1 à 35 000 chez les eucaryotes. La vitesse de synthèse va jusqu’à 50 000 pdb/min pour les eucaryotes et encore plus rapide pour les procaryotes ne présentant pas de chromatine.
L’origine de réplication n’est pas localisée au hasard sur la molécule d’ADN, en effet elle est présente entre petite séquence répétée reconnue par des protéines. Elle mesure environ 200 paires de bases (pdb) pour les procaryotes et 2000 pdb pour les eucaryotes.
Pour l’ADN procaryote, les terminaisons sont doubles pour une origine et sont riches en paires de bases A-T, plus faibles que les paires de bases G-C.
II) Réplication bidirectionnelle
A chaque origine de réplication, il y a formation d’un œil de réplication qui s’agrandit tout le long de l’avancement au niveau des fourches de réplication. Il y a ainsi deux systèmes de réplication qui évoluent en sens opposés. On dit que la réplication est bidirectionnelle.
III) Polymérisation unidirectionnelle & réplication semiconservative
L’ADN bicaténaire est stabilisé par des appariements stricts de bases : d’une part l’adénine (A) et la thymine (T) par 2 liaisons hydrogènes, et d’autre part la guanine (G) et la cytosine (C) par 3 liaisons hydrogènes.
Les deux brins d’ADN sont associés de manière antiparallèle, chacun d’eux possédant une extrémité 5’- phosphate et une extrémité 3’-OH libre.
La polymérisation est unidirectionnelle, en effet les brins étant polarisés la polymérisation se fera toujours dans le même sens : 5’ vers 3’. Il y a formation d’une liaison phosphodiester entre l’extrémité 3’OH du brin en voie d’élongation et l’extrémité 5’phosphate du nucléotide ajouté.
La réplication se fait par copie de l’ADN matriciel. Chaque molécule d’ADN initialement présente se séparera en deux brins d’ADN qui serviront de matrice aux brins néo-synthétisés, il y aura donc un brin parent et un brin fils qui constitueront ensemble la nouvelle molécule d’ADN de la future cellule. La réplication est ainsi semi-conservative.
IV) Réplication semi-discontinue
Nous avons vu précédemment que la synthèse de l’ADN est bidirectionnelle à partir de l’origine de réplication et qu’il y avait formation de fourche de réplication par écartement de deux brins parents. De plus au niveau d’une fourche de réplication les deux brins fils sont synthétisés simultanément.
Nous savons également que la synthèse de l’ADN se fait toujours dans le sens 5’ vers 3’, ceci nécessite donc la présence d’un brin précoce (ou primaire) qui est le brin lu dans le sens de la fourche et d’un brin tardif (ou secondaire) qui est le brin lu dans le sens inverse de la fourche et qui est dit brin discontinu. On parle ainsi de réplication semi-discontinue.
V) Les ADN polymérases
1) Généralités
Les ADN polymérases (ou désoxynucléotidyl-transférase) sont les enzymes responsables de la polymérisation des nucléotides lors de la réplication de l’ADN. Elles sont ADN dépendantes, c’est-à-dire qu’elles ont besoin d’une matrice d’ADN pour produire le brin néo-synthétisé, et pour se faire elles lisent le brin matriciel de 3’ vers 5’ pour synthétiser de l’ADN dans le sens 5’ vers 3’.
Les ADN polymérases procaryotes sont de 3 types (I, II et III) et les ADN polymérases eucaryotes de 5 types (α, β, δ, ε et γ). Dans la suite du cours nous allons prendre en compte les ADN polymérases procaryotes de fonctionnement plus facile.
Les ADN polymérases nécessitent un certain nombre de conditions d’activités :
- Les 4 désoxy-ribo-nucléotides 5’ tri-phosphate (dATP, dTTP, dCTP et dGTP) en quantité équimolaire.
- Des ions magnésiums (Mg2+) qui stabilisent l’ADN et les protéines.
- Une matrice d’ADN (mono ou bicaténaire).
- Une amorce d’ADN ou d’ARN ayant une extrémité 3’OH libre.
2) Activités des ADN polymérases
Lors de la formation de la liaison phosphodiester entre un désoxy-ribo-nucléotides 5’ tri-phosphate et le brin en voie d’élongation, il y a hydrolyse de la fonction triphosphate et formation de pyrophosphate (PPi).
Les ADN polymérases ont des activités bien spécifiques :
- Une activité polymérasique 5’ vers 3’ qui est leur activité principale.
- Une activité exo-nucléasique qui correspond à la dégradation d’une des extrémités du brin néo-synthétisé de l’ADN lors de la réplication et qui peut être de 2 types :
- De 3’ vers 5’, qui correspond à la dégradation à partir de l’extrémité 3’OH. L’activité exo-nucléasique 3’ vers 5’ permet ce qu’on appelle le proofreading, qui correspond à la correction d’un mauvais appariement de base en cassant la liaison phosphodiester et en remplaçant le nucléotide mal apparié.
- De 5’ vers 3’, qui correspond à la dégradation à partir de l’extrémité 5’phosphate, lors de la jonction des segments d’ADN synthétisé sur le brin retardé (cf. suite du cours).
3) ADN polymérases procaryotes dans la réplication
Comme dit précédemment, les ADN polymérases procaryotes sont de 3 types et pour la réplication nous allons principalement étudier le fonctionnement des ADN polymérases I et III.
- Les ADN polymérases I (ou enzyme de Kornberg) sont les plus nombreuses (400 molécules/cellule). Elles présentent l’activité polymérasique 5’ vers 3’ ainsi que les activités exo-nucléasique 5’ vers 3’ et 3’ vers 5’. La vitesse de synthèse des ADN polymérase I est faible (20 nt/s) et ce sont des enzymes peu processive (10-20 nt/évènement) ; ces caractéristiques ne leur permettent pas de faire la majorité de la réplication des ADN procaryotes. Elles sont utilisées dans la réparation de l’ADN ainsi que pour combler les brèches laissées par l’ADN polymérase III.
- Les ADN polymérases III (ou enzyme cœur) sont des multimères hétérogènes de gros poids moléculaire qui sont responsables de la synthèse des fragments longs de l’ADN, ayant une vitesse de synthèse rapide (environ 1000 nt incorporés/s) ainsi qu’une grande processivité (105 nt/évènement). Elles présentent les activités polymérasique 5’ vers 3’ et exo-nucléasique de 3’ vers 5’ mais pas exo-nucléasique de 5’ vers 3’.
VI) Mécanismes de la réplication procaryote chez E-Coli
La synthèse doit respecter certaines propriétés : les deux fourches réplicatives doivent migrer dans des sens opposés, la synthèse de l’ADN se fait dans la direction 5’ vers 3’ et ainsi le brin matriciel est lu de 3’ vers 5’, les deux brins de l’ADN sont antiparallèles et synthétisés simultanément.
1) Les différentes protéines mise en jeu
- Les protéines de reconnaissance reconnaissent les sites d’initiation et de terminaison.
- Les hélicases (ou DNA B) déroulent la double hélice par rupture des liaisons hydrogènes présentes entre les deux brins de l’ADN, avec consommation d’ATP.
- Les protéines SSB (pour single stranded binding protein) ont une forte affinité pour l’ADN simple brin et l’empêche ainsi de se réenrouler lors de la migration des fourches réplicatives.
- La primase est une ARN polymérase ADN dépendante qui synthétise l’amorce.
- Les topo-isomérases relâchent les contraintes de torsion de l’ADN, elles sont de deux types et seule la topo-isomérase de types II consomme de l’ATP. La topo-isomérase de type II d’E-Coli s’appelle l’ADN-gyrase.
- Les ADN ligases (ou DNA G) catalyse la formation de la liaison phosphodiester, mais est incapable de placer les nucléotides. Lors de réparation de l’ADN les nucléotides en place ne sont plus triphosphatés, l’ADN ligase à donc besoin d’un apport en ATP.
2) Origines et terminaisons chez E-Coli
L’origine de réplication possède une séquence répétée de 13 pdb riche en thymine (T), ainsi qu’une séquence GATC présente une dizaine de fois.
Le terminateur mesure environ 350 kb et est composé de 7 séquences quasi identiques de 23 pdb.
3) Les étapes de la réplication procaryote
- Ouverture de la double hélice et formation de la fourche réplicative :L’ouverture de la double hélice sur 40 pdb est permise par reconnaissance de l’origine de réplication par les DNA A. Les topo-isomérases relâchent les contraintes topologiques appliqués à la double hélice par son ouverture.L’ouverture de l’ADN entraîne la formation de l’œil de réplication et des deux fourches de réplication. Les hélicases (DNA B) se mettent alors en place pour permettre le déroulement des deux brins ; les topo-isomérases, elles, sont présentent en aval (en avant) de la fourche permettant d’enlever les contraintes essentiel à l’avancée de l’hélicase. D’autre part les protéines SSB protègent les ADN simples brins pour les empêcher de se réenrouler.
- Elongation du brin précoce dans le sens de déplacement de la fourche :Le brin qui servira de matrice au brin précoce est lu dans le même sens que l’avancée de la fourche, c’est-à-dire de 3’ vers 5’. Au niveau de l’origine de réplication les ADN polymérases nécessitent une amorce qui sera mise en place par les primases. Cette amorce sera ici de l’ARN, l’ADN pouvant être utilisé in vitro. L’ADN polymérase III sera responsable de l’initiation et de l’élongation du brin précoce.
- Elongation du brin tardif dans le sens inverse du déplacement de la fourche :Le brin qui servira de matrice pour le brin tardif doit également être lu dans le sens 3’ vers 5’, mais comme nous l’avons déjà vu précédemment, la fourche se déplace dans le sens inverse et donc l’ADN polymérase III qui sera également responsable de l’élongation du brin tardif. De cette manière sa synthèse sera segmentée en fragment de taille relativement constante à chaque fois que le brin matriciel sera assez « découvert » et ainsi on respectera le sens d’élongation de 5’ vers 3’. Ces fragments sont appelés fragments d’Okasaki.Les fragments d’Okasaki eucaryote mesurent 100 à 200 pdb et les procaryotes 1000 à 2000 pdb. A chaque segment il y a recrutement d’une primase pour la synthèse d’une amorce d’ARN constitué de 10 à 50 nucléotides selon l’espèce.Par la suite les amorces vont être détruites par des protéines à activité ribonucléasique telles que des RNases, et l’ADN polymérase I va compléter la brèche entièrement. La dernière liaison phosphodiester entre l’extrémité 5’ du premier fragment et l’extrémité 3’ du deuxième fragment, ce qui correspond à l’épissage, sera réalisée par la ligase.
- Terminaison :Le terminateur est le site de fixation de protéines « Tus » qui reconnaît les régions Ter. Chez E-Coli, la partie entre les deux terminateurs n’est d’abord pas répliqué, les deux ADN circulaires sont ainsi associés, on utilise alors la topo-isomérase II pour les dissociés. L’ADN polymérase I complètera ensuite les parties non répliquées.
4) Régulation de la réplication chez E-Coli
- Méthylations des séquences GATC au niveau de l’origine de réplication :L’initiation de la réplication nécessite la méthylation des séquences GATC sur les deux brins par la protéine Dam (pour DNA adénine méthylase). L’hémi-méthylation bloque la réinitiation.
- Rôle de la DNA A :L’accumulation de Dna A induit l’initiation de la réplication. Le promoteur de Dna A contient aussi des séquences GATC.
VII) La réplication eucaryote
1) Les ADN polymérases eucaryotes
L’ADN polymérase γ est présente dans les mitochondries mais est codée par un gène nucléaire. Elle est impliquée dans la réplication de l’ADN mitochondrial (16000 pdb) qui code pour 13 protéines, 22 ARNt et 2 ARNr.
L’ADN polymérase α on une fonction de primase. Les ADN polymérases δ et ε sont responsable de la réplication du brin précoce et des fragments d’Okasaki. L’ADN polymérase δ est très processive en présence de PCNA et l’ADN polymérase ε est très processive même en absence de PCNA. Le PCNA (ou proliferating cell nuclear antigen) est une molécule qui augmente fortement la processivité ; elle peut être comparée à un collier coulissant associé à l’ADN polymérase (on parle de clamp β chez E-Coli).
Au niveau du noyau, il y a présence de foyer de réplication de l’ADN, la réplication se fait donc de manière ponctuelle c’est l’ADN qui se déplace et non pas les ADN polymérases.
2) Les télomères
Les chromosomes raccourcissent à chaque division cellulaire. Si l’extrémité des chromosomes était libre il y aurait perte de matériel génétique. Les chromosomes présentent ainsi ce qu’on appelle des télomères (cf. cours de biologie cellulaire) dont leur taille et leur nombre de réplication (qui sont reliés l’un à l’autre) sont très important. En effet lorsque le télomère disparaît, la cellule meurt par apoptose. Le télomère est formé grâce à des télomérases qui sont des ribonucléoprotéines pouvant s’associer à des séquences répétées spécifiques de l’extrémité du chromosome. Les télomérases jouent le rôle de matrice pour les ADN polymérases qui synthétiseront les télomères.
Les télomères ont différents rôles : maintenir l’intégrité des informations génétiques, protéger les ADN vis-à-vis des exo-nucléases, éviter les fusions des chromosomes au niveau des extrémités, rôle dans l’organisation de la chromatine durant l’interphase par interaction avec la membrane nucléaire.
Attention, les télomérases ne sont pas actives dans les cellules différenciées.
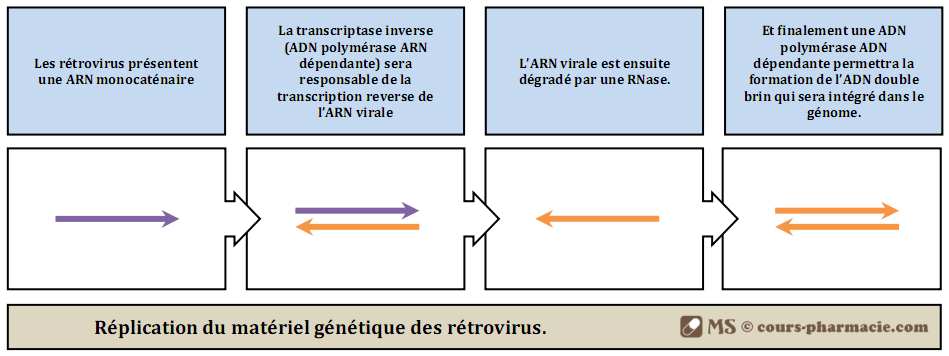
VIII) La réplication du matériel génétique des rétrovirus
Les rétrovirus sont des virus à ARN monocaténaire dont le génome passe au cours du cycle viral, par une intégration sous la forme d’ADN, dans le génome de la cellule hôte. Le plus connu de ces rétrovirus est virus de l’immunodéficience humaine (VIH) ou virus du SIDA.
La réplication du matériel génétique des rétrovirus permet ainsi le passage d’une ARN simple brin à un ADN double brin et ceci grâce à 3 principales enzymes :
- Une ADN polymérase ARN dépendante, qui n’est autre que la transcriptase inverse responsable de la transcription reverse de l’ARN virale. Elle a la caractéristique de synthétiser dans la direction 5′ vers 3′, et nécessite une amorce, une matrice, ainsi que les désoxyribonucléosides triphosphate ; elle ne présente par contre pas d’activité exo-nucléasique 3’ vers 5’.
- Une RNase responsable de la lyse de l’ARN virale.
- Une ADN polymérase ADN dépendante responsable de la formation de l’ADN double brin qui sera intégré dans le génome de la cellule hôte.