- I) Le code génétique
- II) Les acteurs de la traduction
- 1) Les ribosomes
- 2) Les ARNt
- a) Structure des ARNt et ARNt iso-accepteur
- b) Chargement de l’acide aminé sur l’ARNt
- III) Les différentes étapes de la traduction procaryote
- 1) Initiation
- 2) Elongation
- a) Réaction de couplage
- b) Formation de la liaison peptidique et libération du premier ARNt
- c) Translocation
- 3) Terminaison
- IV) Les spécificités de la traduction eucaryote
La traduction correspond au fait que l’ARNm est traduit en protéine : passage de séquences de nucléotides à des séquences d’acides aminés par respect du code génétique. La traduction s’effectue dans le cytoplasme de la cellule.
I) Le code génétique
Le code génétique est un code qui permet la conversion d’une séquence de nucléotides (ADN puis ARN) en séquence d’acides aminés (protéines). Le code implique les bases A, C, T et G ainsi que les 20 acides aminés.
Le code génétique possède différentes caractéristiques :
- Les codons sont des triplets de nucléotides et ils codent pour un acide aminé.
- La séquence du gène et la séquence de la protéine codée sont colinéaires, c’est-à-dire que la longueur du gène et la longueur de la structure primaire de la protéine finale sont proportionnelles.
- Le code génétique est universel. En effet chaque acide aminé dispose d’un ou plusieurs codons et ceci au niveau d’une multitude d’organismes vivants procaryote et eucaryote.
- Le code génétique est redondant (ou dégénéré). Plusieurs codons codent pour un même acide-aminé : on trouve 64 codons et 20 acides aminés. Souvent se sont les deux premiers nucléotides du codon qui définissent l’acide aminé, la redondance est donc due au troisième nucléotide du codon.
- Le code génétique est non-chevauchant. Les nucléotides d’un codon ne participe qu’au code d’un seul acide aminé, ainsi le prochain acide-aminé sera codé par le prochain codon présent sur l’ARNm. On parle du cadre de lecture (ou reading frame).
- Le code possède un système de ponctuation. Le codon d’initiation est le codon AUG (GUG pour ma mitochondrie) et les codons de terminaison sont les codons UAA (ocre), UAG (ambre) et UGA (opale). Le codon UGA (opale) n’est pas présent au niveau de la mitochondrie.
Remarques :
Le cadre de lecture est définit par le codon d’initiation, ainsi le véritable codon de terminaison sera le premier codon qui sera dans le cadre de lecture imposé par ce codon d’initiation.
Parmi les 64 acides aminés, 3 sont des codons de terminaison ou codon stop les 61 restants sont des codons codant.
II) Les acteurs de la traduction
Les acteurs de la traduction sont l’ARN messager (ARNm), les ARN de transfert (ARNt), les ribosomes, les acides aminés, les amino-acyl tRNA synthétases, le Mg2+, le GTP et l’ATP.
1) Les ribosomes
Les ribosomes sont constitués d’ARN ribosomiques (ARNr) et de protéines et sont structurés sous forme de deux sous-unités que ce soit chez les procaryotes ou chez les eucaryotes (cf. également chapitre généralités). Leur taille est définie en unité Svedberg.
- Les ribosomes procaryotes (70S) sont constitués d’une petite sous-unité 30S et d’une grande sous-unité 50S.
- La sous-unité 30S est constituée d’un ARNr 16S (1541 nucléotides) et de 21 protéines.
- La sous-unité 50S est constituée des ARNr 23S (2904 nucléotides) et 5S (120 nucléotides) ainsi que de 32 protéines.
- Les ribosomes eucaryotes (80S) sont constitués d’une petite sous-unité 40S et d’une grande sous-unité 60S.
- La sous-unité 40S est constituée d’un ARNr 18S et de 33 protéines.
- La sous-unité 60S est constituée des ARNr 28S, 5,8S et 5S ainsi que de 49 protéines.
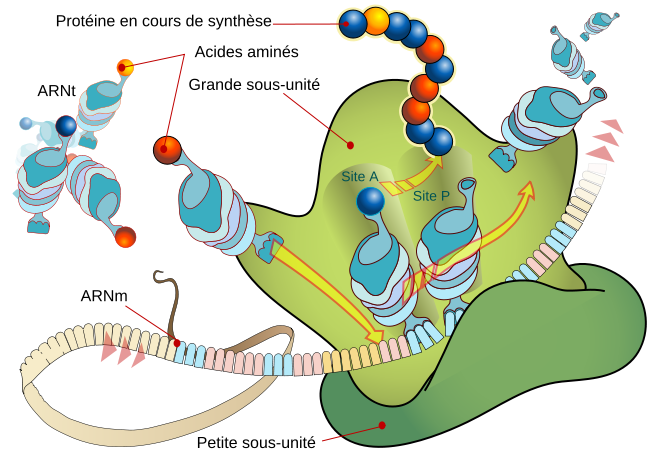
Topographie schématique du ribosome bactérien :
Le ribosome bactérien comporte des sites spécifiques :
- Site A : (= site Acide-aminé ou Accepteur) fixation des acides aminés.
- Site P : (= site Peptidique ou Donneur) fixation de f-Met.
- Site E : (= site Exit) sortie de l’ARN de transfert.
- Site EF-G : présent au niveau de la grande sous-unité.
- Site EF-Tu : présent au niveau de la petite sous-unité.
Attention chez les eucaryotes le premier acide aminé est la méthionine et non pas la f-Met présent chez les procaryotes.
L’enchaînement des ribosomes sur l’ARNm forme le polysome, il permet d’augmenter l’efficacité de la traduction. La distance minimale qui sépare deux ribosomes est de 100 nucléotides.
Au niveau des ribosomes associés au réticulum endoplasmique les protéines en voie de synthèse pénètrent dans les vésicules du réticulum directement après le site E (cf. cours de biologie cellulaire).
2) Les ARNt
a) Structure des ARNt et ARNt iso-accepteur :
Les ARNt ont une structure secondaire en forme de trèfle à 3 feuilles et une structure tertiaire en forme de L à l’envers. Lors du mécanisme de traduction il y a un appariement antiparallèle entre l’ARNm et l’ARNt : reconnaissance codon-anticodon au niveau de la boucle de l’anticodon.
Il existe une flexibilité dans l’appariement des bases en position 3 du codon et en position 1 de l’anticodon, cette flexibilité s’appelle le wobble.
Remarque : I est une base purique qui peut s’apparier avec U, C et A mais pas G.
Les ARNt possèdent également un bras de l’acide aminé qui le fixe en 3’ (CCA) sur le ribose, il s’agit d’une liaison covalente : liaison ester riche en énergie. Les acides aminés ne vont ainsi par arriver libre sur le ribosome mais associés à leurs ARNt respectifs. On trouve 40 à 60 ARNt différents par cellule, il existe donc plusieurs ARNt différents pour un acide aminé, on les appelle ARNt iso-accepteurs. (Ecriture : tARN leu 1)
b) Chargement de l’acide aminé sur l’ARNt :
La formation du complexe amino-acyl-tARN (aa-tARN) nécessite une Amino-acyl-tRNA-synthétase spécifique de l’acide aminé, qui doit ainsi reconnaître toutes les formes de codon de cet acide aminé. Le chargement correct de l’ARNt est un élément important dans la fidélité de la traduction.
L’acide aminé (aa) est tout d’abord activé et cette activation nécessite de l’énergie sous forme d’ATP pour permettre la formation d’aa-AMP (liaison anhydride mixte).
La liaison formée entre l’ARNt et l’acide aminé est une liaison covalente de type carboxy-ester. Les Amino-acyl-tRNA-synthétase sont au nombre de 20 dans la cellule, autant qu’il y a d’acides aminés qui rentrent en compte dans la traduction. L’acide aminé complexé peut ainsi s’associer à la chaîne.
III) Les différentes étapes de la traduction procaryote
1) Initiation
Un ribosome reconnaît le début de la séquence codante, il utilise des signaux d’adressage en amont entre -8 et-13 du codon initiateur (AUG) qui correspond à la séquence de Shine-Dalgarno ou RBS (AGGAGG). Il y a appariement antiparallèle de bases entre l’ARNm et la petite sous-unité (30S) du ribosome, dû à une complémentarité de séquences entre l’ARNm et l’ARNr 16S.
Les bactéries nécessitent un acide aminé particulier pour l’initiation ; cet acide aminé est la méthionine et elle nécessite une formylation sur l’extrémité NH2 (ajout d’un formyl) pour former la f-Met, c’est un phénomène pré-traductionnel. Cette formylation est réalisée par un cofacteur, la vitamine B9 (ou tétrahydrofolate) qui reconnaît l’ARNt caractéristique et responsable du transport de la f-Met. La particularité de conformation de cet ARNt lui permet d’être placé directement dans le site P et non pas dans le site A.
L’initiation est permise grâce à la présence de facteurs d’initiation (IF pour Initiation Factor) :
- IF 1 est le facteur de dissociation du ribosome 70S.
- IF 2 est un facteur assurant la fidélité de reconnaissance entre l’ARNt et l’acide aminé. Il possède également une activité GTPasique (c’est-à-dire d’hydrolyse du GTP).
- IF 3 est un facteur nécessaire à la fixation spécifique de 30S sur l’ARNm et de contrôle de l’équilibre entre la forme associé et dissocié du ribosome (facteur anti-réassociation).
Par la suite le complexe 70S est reformé : lorsque l’ARNt fixé à la formyl-méthionine est fixé à la petite sous-unité de l’ARNr, il y a hydrolyse du GTP et la grande SU se fixe sur le complexe.
2) Elongation
L’élongation correspond à une synthèse protéique par ajout d’acides aminés à l’extrémité C-Terminale de la chaîne peptidique naissante, réaction catalysée par l’activité peptidyl-transférase de la grande SU des ribosomes. La lecture de l’ARNm par le ribosome se fait de 5’ vers 3’. Il y a formation d’une liaison amide particulière appelée liaison peptidique, les deux fonctions formant la liaison étant portées par le carbone-α de deux acides aminés différents. Cette réaction entraîne l’élimination d’une molécule d’eau.
L’élongation également est permise par la présence de facteurs d’élongation (EF pour Elongation Factor) : EF-Tu ; EF-Ts et EF-G.
Pour chaque liaison peptidique formée on peut caractériser 3 étapes : la réaction de couplage, la formation de la liaison peptidique et la translocation.
a) Réaction de couplage :
L’étape de couplage correspond au transfert de l’acide aminé complexé à l’ARNt sur la chaîne protéique en voie d’élongation. On peut faire la remarque que durant la traduction c’est l’extrémité N-terminal (fonction amine) qui sort en premier du ribosome. Ainsi c’est l’extrémité C-terminale (fonction carboxyle) du premier acide-aminé qui permettra la formation de la liaison peptidique avec la fonction amine du deuxième acide-aminé. Ainsi le deuxième complexe aa-ARNt arrive dans le site A, la f-Met étant positionnée dans le site P.
b) Formation de la liaison peptidique et libération du premier ARNt :
La liaison riche en énergie qui lie le premier ARNt avec la f-Met se rompt amenant l’énergie pour permettre la formation de la liaison peptidique, ceci étant uniquement possible à ce moment là car la fonction COOH était jusqu’alors engagée dans la liaison à l’ARNt. L’ARNt est alors expulsé vers le cytoplasme où il sera recyclé, en même temps que la formation de la liaison peptidique, réaction catalysée par l’activité peptidyl-transférase de la grande sous-unité.
c) Translocation :
Comme dit précédemment la lecture de l’ARNm se fait de 5’ vers 3’, de ce fait le ribosome se déplace de 3 nucléotides (ou d’un codon) dans cette direction de telle sorte que l’ARNt portant les deux premiers nucléotides se retrouve dans le site P, il y a translocation. L’ARNt portant le troisième peut ensuite prendre place dans le site A à nouveau libre.
Ce cycle de trois étapes va donc se reproduite autant de fois qu’il est nécessaire jusqu’au codon stop.
L’ATP apporte deux liaisons riches en énergie, et est consommé lors de l’activation de l’acide aminé, c’est-à-dire lors de la formation d’aa-AMP qui sera ensuite complexé par l’amino-acyl-tRNA-synthétase à l’ARNt correspondant. Il y aura ainsi une liaison riche en énergie entre l’acide aminé et son ARNt. Mais comme énoncé précédemment l’énergie utilisée pour la formation de la liaison peptidique est l’énergie récupérée par la rupture de la liaison riche en énergie du complexe aa-ARNt précédent et non pas de la liaison du complexe s’ajoutant à la chaîne peptidique en cours d’élongation.
Les facteurs d’élongation font partie de la famille des protéines G. Les protéines G sont des protéines ayant la propriété de n’être actives que lorsqu’elles sont liées au GTP. Le GTP est par la suite hydrolysé en GDP et Pi, par l’activité GTPasique de ces protéines G.
- Le premier GTP est utilisé lors du positionnement du complexe aa-ARNt dans la loge A du ribosome. En effet l’acide aminé-ARNt forme un complexe avec EF-Tu, qui lui permet de se fixer sur le ribosome (site A). Après fixation du complexe, le GTP est hydrolysé en GDP et le facteur EF-Tu-GDP se détache.
- Le deuxième GTP est utilisé lors de la translocation, également par un facteur d’élongation ayant une activité GTPasique sur le même principe que précédemment.
3) Terminaison
La terminaison de la traduction se fait au niveau des codons stop UAA, UAG et UGA qui ne codent pour aucun acide aminé. Ces codons stop sont reconnus par les facteurs de terminaison RF 1, RF 2 et RF 3 (RF pour Releasing Factor) :
- RF 1 reconnaît UAA et UAG.
- RF 2 reconnaît UAA et UGA.
- RF 3 stimule l’activité des 2 autres facteurs.
La liaison ester unissant l’ARNt au dernier acide aminé de la chaîne peptidique est hydrolysée par la peptidyl-transférase. Le ribosome se redissocie en deux sous-unités qui pourront recommencer de nouvelles lectures d’ARNm.
La terminaison fait intervenir, tout comme l’initiation, l’hydrolyse d’une molécule de GTP.
Remarque :
La traduction bactérienne peut être inhibée par des antibiotiques tels que les aminosides qui inhibent la petite sous-unité ou les macrolides qui agissent au niveau de la grande sous-unité.
Vidéo résumant les différents mécanismes entrant en jeu dans la traduction.
IV) Les spécificités de la traduction eucaryote
Le ribosome est de taille différente et composé d’ARN ribosomiques différents bien que la structure générale et l’activité soit comparable (cf. début du cours).
Le codon initiateur est également AUG et c’est généralement le 1er AUG présent sur l’ARNm. On peut trouver le triplet ATG qui donnera le codon AUG sur l’ADN-sens au niveau de la séquence de Kozak (GCCGCC(A/G)CCATGG).
Comme dit précédemment, chez les eucaryotes le premier acide aminé est la méthionine et non pas la f-Met présent chez les procaryotes. La méthionine sera le plus souvent enlevée juste après la synthèse de la chaîne peptidique.
Les facteurs d’initiation sont du type eIF (pour eukaryotic Initiation Factor), d’eIF1 à eIF6.
Les facteurs d’élongation sont également du type EF (EF1α, EF1β et EF2).
Les facteurs de terminaison sont du type eRF (pour eukaryotic Releasing Factor).